在哺乳动物发育过程中,成熟单倍体配子的形成起始于原始生殖细胞(Primordial Germ Cells,PGCs)的谱系发育。以小鼠为模型的研究表明,PGCs从胚胎期形成,历经迁移至生殖嵴直至完成性别分化的整个过程中,始终维持着高度动态的表观遗传重编程状态。这一特征性现象主要表现为全基因组范围的DNA去甲基化[1]、H3K9me2组蛋白修饰的显著减少以及H3K27me3修饰的广泛获得[2]。现有证据表明,这种大规模的表观遗传重塑可能对维持PGCs的多能性潜能具有关键作用[3]。
随着性别分化完成,雌性生殖细胞在胚胎发育第13.5天(E13.5)开始有序进入减数分裂程序。至出生前,绝大多数雌性生殖细胞发育并停滞在第一次减数分裂前期的双线期(diplotene stage)。这一复杂的发育进程涉及DNA复制、程序性DNA双链断裂的形成与修复、同源染色体联会以及交叉互换等关键生物学事件,这些机制共同确保了遗传物质的稳定传递和遗传多样性。值得注意的是,在减数分裂前期进展过程中,很可能伴随着特定的表观遗传重编程事件,这些事件可能通过调控减数分裂相关基因的表达网络来精确协调减数分裂进程。基于上述背景,本研究重点关注以下科学问题:(1)在第一次减数分裂前期,哪些特定的表观遗传修饰发生了显著的重编程变化?(2)这些表观遗传修饰如何影响减数分裂关键基因的表达调控?
2025年7月12日,中国科学院动物研究所王震波研究员团队与广东省第二人民医院生殖中心团队合作,在《Nucleic Acids Research》发表了题为"Re-establishment of H3K9me2 eliminates the transcriptional inhibition of ST18 on meiotic genes and orchestrates female germ cell development"的研究论文。该研究首次系统阐明了H3K9me2表观遗传修饰在雌性生殖细胞减数分裂进程中的关键作用,为理解减数分裂调控机制及女性生殖发育障碍提供了重要理论突破。
研究团队发现,在雌性生殖细胞发育过程中,H3K9me2修饰的动态重建对减数分裂的顺利进行至关重要。这一过程主要依赖于组蛋白甲基转移酶EHMT1的表达上调,其介导的H3K9me2修饰可有效抑制转录抑制因子ST18的表达。在EHMT1缺失小鼠中,H3K9me2重建失败导致ST18异常激活,进而抑制多个关键减数分裂基因(如Stra8、Sycp3、Dmc1等)的转录,最终引发减数分裂前期阻滞及生殖细胞凋亡。该研究揭示了H3K9me2(EHMT1)-ST18-Meiotic genes调控轴在减数分裂基因表达中的重要作用,为深入理解女性生殖细胞发育障碍的分子机制提供了新的研究方向,并为相关生殖疾病的临床干预策略提供了潜在靶点。
该研究首先通过免疫荧光染色技术系统分析了雌性生殖细胞在不同发育阶段(E11.5有丝分裂期、E13.5有丝-减数分裂转换期、E15.5减数分裂期)的组蛋白修饰动态变化,发现H3K9me2修饰在减数分裂进程中呈现显著重建和持续累积的趋势。进一步利用Cut&Tag-Seq技术对H3K9me2在全基因组的分布进行精确分析,证实其信号密度在减数分裂过程中逐步增强,提示该修饰可能参与调控减数分裂基因表达和减数分裂进程。为探究H3K9me2重建的分子机制,研究团队构建了生殖细胞EHMT1条件性敲除小鼠模型。结果显示,EHMT1缺失导致雌性小鼠不育,其生殖细胞因减数分裂阻滞而大量凋亡。深入分析发现,EHMT1缺失小鼠的生殖细胞存在DNA双链断裂修复缺陷、转录组紊乱,且多个减数分裂关键基因(如Dmc1、Spata22、Meiob、Sycp1、Scye3、Msh4、Msh5、Mei1等)表达显著下调。同时,H3K9me2修饰的重建完全受阻。结合Cut&Tag-Seq与转录组联合分析,研究人员鉴定出一组在EHMT1缺失条件下异常上调的转录抑制因子,包括ST18、ZNF462、KIF12和ETV6。通过双荧光素酶报告基因实验,研究团队进一步证实ST18可直接结合并抑制部分减数分裂基因的转录,而H3K9me2的重建可有效解除ST18的抑制作用,从而确保减数分裂程序的正常进行。
中国科学院动物研究所王震波研究员为该研究的通讯作者;中国科学院动物研究所博士生赵丙旺、已毕业硕士张颐娜和广东省第二人民医院生殖中心的孟铁刚研究员为共同第一作者;中国科学院动物研究所的博士生徐源鸿、鲁宜珂、孙思敏、郭佳妮和杨雪梅对本研究也做出了重要贡献。该研究得到了国家重点研发计划(2022YFA0806302)、国家自然科学基金(82171646)和北京市自然科学基金(Z240020)的资助。感谢器官再生与智造全国重点实验室提供的技术平台与资源保障。
原文链接:https://academic.oup.com/nar/article/53/13/gkaf657/8198050
[1] Zeng Y,Chen TP. DNA methylation reprogramming during mammalian development. Genes Dev. 2019;10:257.
[2] Seki Y,Hayashi K,Itoh K et al..Extensive and orderly reprogramming of genome-wide chromatin modifications associated with specification and early development of germ cells in mice. Dev Biol. 2005;278:440–58.10.1016/j.ydbio.2004.11.025.
[3] Seki Y,Yamaji M,Yabuta Y et al..Cellular dynamics associated with the genome-wide epigenetic reprogramming in migrating primordial germ cells in mice. Development. 2007;134:2627–38.10.1242/dev.005611.
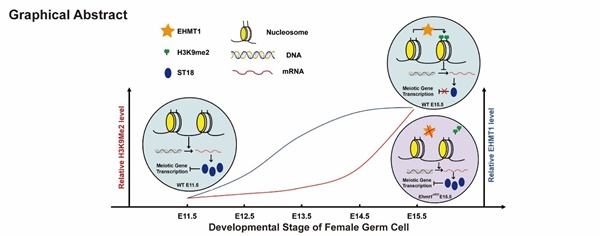
图1:本研究的图片摘要